东亚人群毛干蛋白中单氨基酸多态性检测方法建立与个体识别应用*
来源:优秀文章 发布时间:2022-12-02 点击:
吴佳蕾 季安全 丁冬升 丰 蕾**叶 健**
(1)中国人民公安大学研究生院,北京 100038;
2)公安部物证鉴定中心,现场物证溯源技术国家工程实验室,法医遗传学公安部重点实验室,北京 100038)
近年来,随着质谱技术的不断发展,蛋白质组学进入了高速发展的时期,并且获得了丰硕的成果,依据质谱技术的蛋白质组学被认为是解决众多生物学问题的有效手段[1]。基因组测序的研究成果逐渐积累,蛋白质序列数据库不断增加[2-4],生物信息学相关的分析工具日渐成熟[5],促使分析不同个体蛋白质组中的遗传差异如单氨基酸多态性(single amino acid polymorphism,SAP)成为可能。
在法庭科学领域中,来源于人体的毛发是案件现场最为常见的生物物证之一,毛发分为毛囊和毛干两部分,毛囊中含有细胞核基因组DNA,可采用现有的短串联重复序列(short tandem repeat,STR)检验方法进行DNA个体识别[6],毛干中无细胞形态,核DNA已高度降解,无法使用现有的STR检验方法。然而,大部分案件现场提取到的毛发物证并不带有毛囊,单独的毛干至今缺少有效的个体识别方法。目前,法庭科学对毛干的检测主要有两种方式:一种是运用显微形态检验对毛发的外观进行观察比对,该方法需要主观经验的判断,缺乏科学的统计分析理论和基础,其检验结果在实际案件中的应用面临巨大的挑战[7],在2009年美国国家科学院关于法医学的报告《加强美国法医学:前进之路》中被认为“非常不可靠”,之后进一步调查甚至发现因毛发显微形态结果的错误陈述导致错案的发生;
另一种对毛干物证的检测方法是通过测序检测线粒体DNA的2个高变区[8-9],得到单核苷酸多态性(single nucleotide polymorphism,SNP)的差异,由于线粒体是母系遗传,因此该检测具有母系遗传的特点,存在识别率不高、具有异质性、只能排除不能认定等缺点,无法做到个体识别。尝试利用蛋白质组学技术对人体毛干中的蛋白质进行检测,从而获得具有个体识别潜力的SAP位点,成为解决毛干个体识别难题的新途径。SAP位点检测是利用毛干蛋白进行个体识别的重要前提条件。根据中心法则,每个SAP位点都在DNA上有对应的非同义SNP(non-synonymy SNP,nsSNP)位点,可用SNP位点在东亚人群的频率、通过乘法法则进行个体识别能力的计算[10-11]。
本文对6名成年健康志愿者毛干样本使用离子液体法提取毛干蛋白质组、质谱检测,提取与检测独立重复两次,分析毛干样本中蛋白质组成,而且通过自建的东亚人群SAP蛋白序列数据库分析鉴定每名个体的SAP分型,阐明了不同个体毛干中的SAP差异性。
1.1 毛干蛋白质组的离子液体提取
本文中所用人体生物样本来源于志愿者捐赠,收集了6名中国汉族无关健康个体自然脱落头发和口腔拭子,其中男女各半。样本采集工作通过了公安部物证鉴定中心伦理委员会的伦理审查,并且征得了各志愿者的知情同意。每个个体取2根头发,去除毛囊及发根,剪取整根头发的近发根端2 cm作为分析样本。
离子液体C12Im-Cl可以破坏蛋白质中的氢键网络,对于不同组织中的蛋白质具有很好的溶解性。取2 cm毛干单根样本使用50%乙醇/水溶液清洗两次,用于去除头发表面油脂及污染物。将清洗后的毛干取出并剪碎至约1~2 mm,加入100 μl裂解液中(0.1 mol/L Tris(2-carboxyethyl)phosphine(TCEP,SIGMA),10% C12Im-Cl(m/v)溶 于0.1 mol/L Tris,pH 7.6)[12-13],水浴超声20 min后,放入振荡器内继续37℃振荡过夜,而后取出并用细胞超声破碎仪破碎匀浆至毛干可溶解至肉眼不可见。将毛干蛋白溶液放入95℃水浴5 min后,放入高速离心机16 000×g离心40 min。取上清液至入FASP膜离心管内(10 k,Sartorius AG),16 000×g离 心30 min,后 用ABC溶 液(50 mmol/L NH4HCO3,pH8.0)清 洗,清 洗 完 成 后 加 入30 mmol/L碘 代 乙 酰 胺(iodoacetamide,IAA,SIGMA)的ABC溶 液 中 避 光 反 应20 min,后16 000×g离心20 min。离心完成后加入ABC溶液清洗3次。更换FASP膜下衬管。在膜上加入2 μl胰蛋白酶(2.5 g/L),37℃水浴4 h,再次加入2 μl胰蛋白酶(2.5 g/L),37℃水浴12 h。酶解完成后16 000×g离心20 min,并用Qubit定量(蛋白质定量试剂盒,Invitrogen,Thermo Fisher)所得肽段。
1.2 质谱检测
质谱数据由Thermo Easy-nLC 1000液相色谱和Q-Exactive组合型四极杆Orbitrap质谱仪联用检测获取,上样量为1 μg。
Nano RPLC的色谱分离条件:流动相A为98%H2O+2%ACN+0.1%FA(均为体积分数);
流动相B为98%ACN+2% H2O+0.1% FA(均为体积分数);
首先将10 μl 100%的A上样至C18预柱(3 cm×0.15 mm),压力为300 Bar,然后在C18毛细管柱(15 cm×0.1 mm)上以600 nl/min的流速分离肽段,梯度如下:2%B(0 min)-5%B(0.1 min)-23%B(55 min)-40% B(70 min)-80% B(72 min)-80% B(85 min)。Q-Exactive的质谱参数:正离子模式,特征肽段参数的选择:采集方式为全扫描/数据依赖二级扫描(Full MS/DD-MS2,TOPN),一级扫描范围为m/z300~1 800,一级扫描分辨率为70 000,自动增益控制(AGC)为1×106,离子最大累积时间为60 ms;
二级扫描分辨率为17 500,AGC为5×105,离子最大累积时间为60 ms,TOPN=20(前20强),隔离窗口设为m/z2,碰撞能(NCE)为28。
1.3 东亚人群SAP蛋白质序列数据库构建
根据AnnoVar软件[14-15](2019Oct24)中hg19基因组中编码基因的Ensembl注释,获得包含全部参考型SAP的蛋白质序列。蛋白质编码区域的nsSNP变异信息来源于ExAC数据库(http://exac.broadinstitute.org),保留东亚人群中突变频率高于0.1%的nsSNP,每个nsSNP对应1条含有突变型SAP的蛋白质序列。合并参考蛋白质序列和突变蛋白质序列数据,获得东亚人群SAP蛋白质序列数据库。
该数据库包含nsSNP基因组中的位置、分型和在东亚人群中的基因频率、对应的SAP分型、SAP所在的蛋白质,共包含60 551个蛋白质上的25万个SAP位点。
1.4 数据库的搜库与SAP鉴定
人全蛋白质数据库搜索:质谱检测所得质谱数据文件(*.raw)采用pFind Studio(版本3.1)进行数据库检索[5]。人全蛋白质数据库下载于UniProt,版本为proteome_UP000005640,共包含74 470个蛋白质序列,采用反库控制结果的假阳性率(FDR)。pFind软件搜库设置为3个漏切,全酶切,前体离子允许质量偏差为±10 ppm,碎片离子允许质量偏差为±20 ppm,FDR≤1%,Open Search不勾选。半胱氨酸氨基甲基化修饰(carbamidomethyl[C])为固定修饰,蛋白质N端乙酰化(acetyl[PronteinN-term])和甲硫氨酸氧化(oxidation[M])修饰为可变修饰。
东亚人群SAP蛋白质序列数据库搜索:首先提取6个个体外显子测序获得的全部nsSNP,使用AnnoVar注释获得对应的SAP信息,并加入到自建SAP蛋白质序列数据库中形成新的库。使用pFind对新库进行搜库,参数设置同上,利用自建的数据分析流程从全部特异性肽段获得含SAP肽段,提取SAP位点信息,并根据建库时的SAP与nsSNP对应注释表,获得鉴定到的SAP位点信息、对应的SNP位点信息以及SNP位点在人群中的突变发生频率。根据SAP与标准hg19基因组编码SAP一致与否,分类为参考型和突变型。
1.5 外显子测序
上述6个个体口腔拭子提取全基因组DNA,经NanoDrop 2000定 量 取500 ng,浓 度≥5 μg/L DNA,委托艾吉泰康生物科技(北京)有限公司进行全外显子测序。全外显子组测序(whole exome sequencing,WES)利用液相探针富集外显子区域DNA序列,检测覆盖区域为58 Mb,测序深度≥100×,测序数据量≥9 G。
1.6 随机匹配概率计算
SAP位点对应至相应的SNP位点后,对SNP位点的选择和计算采用以下原则:a.突变型SNP位点基因频率≥0.1%;
b.非同一染色体上的SNP位点为独立遗传;
c.同一染色体上距离>2×105bp假设遗传独立;
d.当两个或以上位点距离在2×105bp内时,取频率最低SNP位点用于计算。随机匹配概率(random matching probability,RMP)的计算使用乘法原则。以SNP不同分型在东亚人群中统计频率作为等位基因频率,假设突变型等位基因频率为p,参考型等位基因等位基因频率为q,则突变型纯合子的基因型频率为p2,参考型纯合子的基因型频率为q2,杂合子的基因型频率为2pq,乘积各位点的基因型频率计算个体随机匹配概率。RMP=基因型频率(SNP 1)×基因型频率(SNP 2)×基因型频率(SNP 3)……。
统计分析作图软件为Graph Pad。
2.1 毛干蛋白质检出情况
使用离子液体前处理方法,2 cm毛干酶解后肽段为(48.19±10.12)μg(n=12,中值=49.36 μg)。来源于6个个体的12个毛干样本,分两个批次进行蛋白质提取与质谱检测,A组为第一批次检验样本,B组为第二批次检验样本。A组6个样本的肽段检出为1 826~2 671个(2 180±345),蛋白质检出数量为216~400个(284±71),特异性肽段检出数量为771~1 012个(885±112)。B组6个样本的肽段检出为1 406~2 524个(1 874±389),蛋白质检出数量为212~366个(267±68),特异性肽段检出数量为569~951个(744±128)。两批次12个样本共检出肽段数量为1 406~2 671个(2 027±385),蛋白质数量为212~400个(276±67)。特异性肽段数量为569~1 012个(814±136)。各样本的检出结果详见表1。为分析毛干中蛋白质细胞组成功能,进行GO分析。对A、B两组中组成成分中最多的前5类蛋白质作图,发现组成毛干最多的5类蛋白质分别为:细胞外泌体蛋白、角蛋白丝蛋白、中间丝蛋白、髓鞘蛋白、细胞外基质蛋白(图1)。由于角蛋白(keratin)和角蛋白相关蛋白(keratinassociated protein,KAP)是毛发的重要组成部分,单独对其进行分析。全部12个样本中,角蛋白和角蛋白相关蛋白共占所有检测到蛋白质种类的25%~44%(图2a),其中每个个体检出的角蛋白有40~51种,角蛋白相关蛋白41~51种(图2b)。12个样本中共检出52种角蛋白,其中有32种角蛋白在所有样本均检出,占61.5%;
共检出KAP 58种,所有样本均有检出的KAP为30种,占51.7%。具体的角蛋白及KAP检出情况见表S1和S2。

Fig.1 The main cellular components of the proteins detected in hair shaft
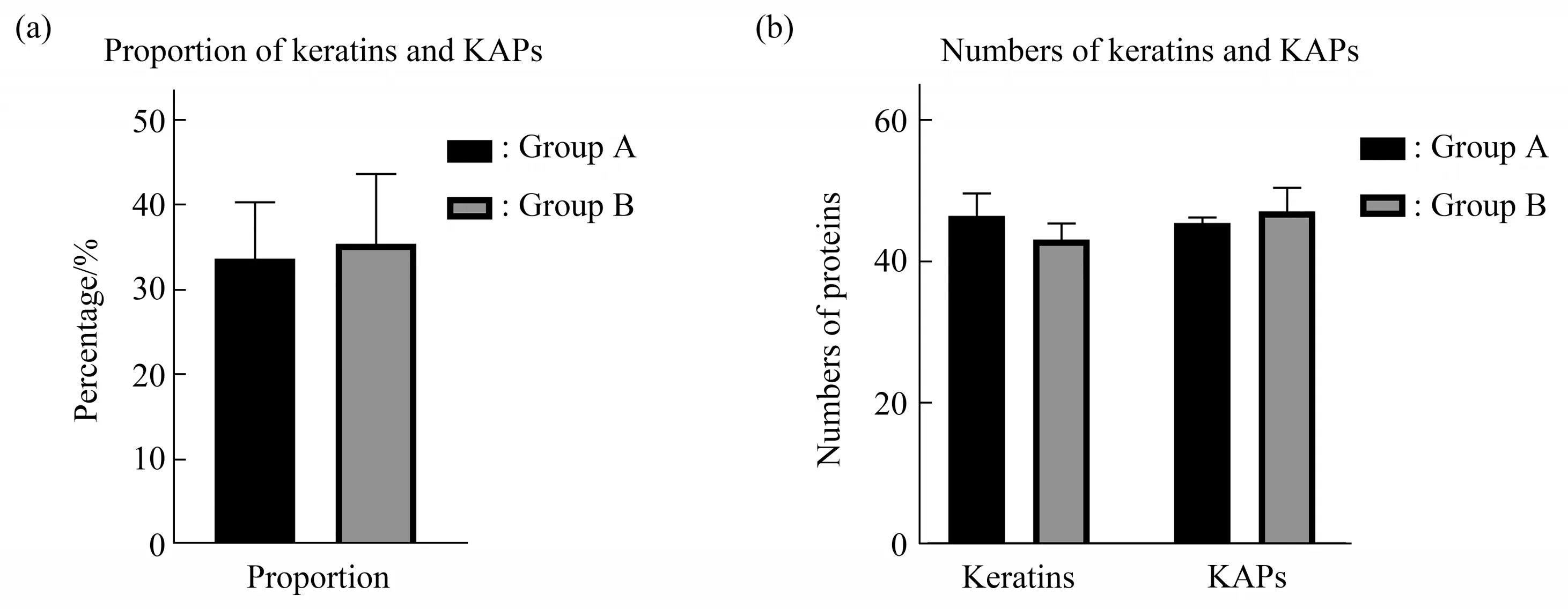
Fig.2 The proportion and numbers for keratins and KAPs detected in hair shaft
为进一步探讨批次间对于检测结果的差异,对A、B两个批次的实验结果进行配对t检验,检出肽段(P=0.24)、蛋白质(P=0.75)显示批次间均无显著性差异。两个批次检测到最多的前5类蛋白质是一致的,说明建立的离子液体毛干蛋白质组质谱检测方法稳定性良好。同时,对A、B两组的角蛋白和角蛋白相关蛋白在该样本所有检出蛋白质中的占比、角蛋白检出数量和角蛋白相关蛋白检出数量进行配对t检验(P=0.75;
P=0.80),发现两组均没有显著性差异。
为分析同一个体蛋白质检测重现性,对同一个人A、B两批次的检出蛋白质种类进行比较,蛋白质检出重复率分别为54.7%、61.3%、63.5%、60.7%、67.8%和67.6%(图3a)。对样本F202A和F202B分别进行两次质谱技术重复,蛋白质检出重复率分别为64.4%和66.2%。通过比较质谱技术重复和样本重复检出蛋白质的重复率,发现除样本F3相差略大,其他5个个体的两批次检出蛋白质重复率与质谱重复的检出蛋白质重复率接近。对同一个体两批次合并后作为一个样本,分析不同样本的检测蛋白质一致性的累积交集与累积并集(图3b),发现随着样本的增加共检出(累积并集)的蛋白质数量呈上升趋势,均检出(累积交集)的蛋白质数量呈下降趋势,其中6个样本共检出蛋白质731个,均检出蛋白质175个。

Fig.3 Protein groups identified in hair shafts from 6 individuals
2.2 SAP位点鉴定
对搜库软件输出的全部特异性肽段进行序列分析,提取SAP分型,与标准基因组hg19编码SAP相比,相同的为参考型SAP,不同的为突变型SAP。结果显示,不仅可以检测到突变型SAP或参考型SAP,也可同时检测到两种分型。如在样本M2A中,同时检测到Sialidase-2蛋白上第41位氨基酸的两种SAP分型,参考型SAP(R),位于肽段IPALLYLPGQQSLLAFAEQR(图4a)和突变型SAP(Q)位于肽段IPALLYLPGQQSLLAFAEQQASK(图4b)。根据建数据库时的SAP与nsSNP对应的注释表,推导出相应的nsSNP分型,即蛋白质谱检测到的nsSNP_pro,与外显子测序获得的nsSNP分型进行比较分析,其中一致的SAP为validated SAP。
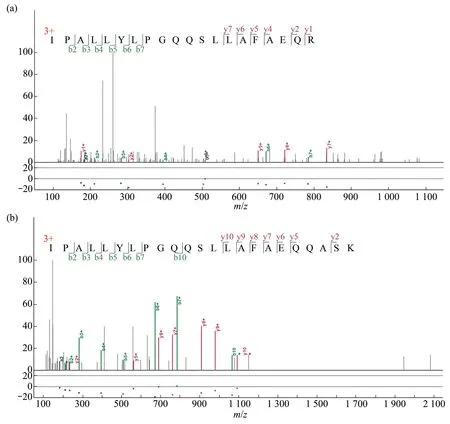
Fig.4 Fragment mass spectrogram of peptides including SAP
从6个个体的12样本中共计鉴定到321个SAP,平均每个样本鉴定到(132±17)个SAP,包含(19±4)个突变型和(113±14)个参考型(表2)。其中A组每个样本鉴定到的SAP位点数量分别为(137±16)个,B组为(126±18)个。A组validated SAP为(127±13)个,B组为(118±17)个。经分组t检验显示,A、B两组检出的全部SAP、突变型和参考型SAP数量无显著性差异(P>0.05)。所有SAP的检出详细信息见表S3。
为比较各样本SAP分型的差异,去除全部检测一致的参考型SAP位点后,仅对存在分型差异的SAP位点(即在任一样本中检出突变型SAP)进行了汇总(图5),共计72个SAP位点。其中有10个位点在所有12个样本中均有检出,对应的nsSNP在东亚人群中的频率分布从0.008到0.353 5。大于0.005的等位基因称为常见等位基因(common allele),在群体中稳定遗传且存在显著的个体差异性。该10个nsSNP位点均为常见等位基因,可作为个体差异性位点。
对同一个体两个样本中检出的大部分位点保持了较好的一致性,也有个别样本存在差异。例如对于rs2071560相应的SAP,F3、F203、M1、M2和M3的两个批次样本检出的分型都是一致的,但F202的B样本检出了杂合分型,而A样本只检测到了突变型。
对12个样本的nsSNP_pro与外显子测序nsSNP结果比较,统计结果为:a.完全匹配占67%,即nsSNP_pro与nsSNP完全一致,包括突变(标黑)、杂合(标橙)、参考分型(标蓝);
b.半匹配占27%,即nsSNP为杂合型,而nsSNP_pro只检测到其中一种分型,漏检了另一种分型(标黄);
c.错误匹配占6%,即nsSNP_pro检出了nsSNP不存在的分型(标绿),如nsSNP为参考型纯合,而nsSNP_pro检出了突变型,或者nsSNP为突变型纯合,而nsSNP_pro检出了参考型(图5)。
2.3 个体识别随机匹配概率结果
为评估获得的SAP位点对于个体识别的区分能力,将SAP对应nsSNP的基因频率用于随机匹配概率的计算。出于准确性考虑,仅使用全外验证准确的validated SAP位点计算RMP(表3)。A组RMP为3.5×10-3~1.0×10-9,中值为1.1×10-4;
B组RMP为1.4×10-2~1.5×10-6,中值为1.6×10-5。经t检验结果显示,A、B两组RMP没有显著性差异(P>0.05)。将每个志愿者A、B两批次检出的SAP合并后计算RMP,较单批次的结果降低1~2个数量级,中值达到1.3×10-6。当使用10个在12个样本中均检出的SAP(图5中的TOP10)进行RMP的估算时,F3、F202、F203、M1、M2、M3的RMP分别为3.4×10-1、9.9×10-3、8.0×10-2、2.0×10-4、7.2×10-2、1.6×10-3。
假定半匹配和错误匹配是随机的,那么理论上来说,如果一个人毛干检测获得的nsSNP_pro,与同一个人或其他无关个体的外显子nsSNP验证比较,来源于同一个体时得到的validated nsSNP_pro(self)数量最多,相应计算RMP(self)值也最低。基于以上假设,本文尝试将每个人的nsSNP_pro结果与其他5个人的测序结果匹配后分别计算RMP(other)。结果显示,当蛋白质与不同的DNA进行匹配时,来源于同一个人的蛋白质和DNA检出的validated nsSNP_pro数量最多。除样本F3以外,基于validated nsSNP_pro(self)计算的RMP(self)也最低,且RMP(self)值越低,与其他个体DNA匹配计算得到的RMP(other)的差距就越大,即个体区分能力越高,最多可相差6个数量级(M3样本)(表4)。对于样本F3,由于检测获得的SAP位点数量偏少,计算RMP(self)值仅为10-4,尽管RMP(self)值并不是最低,但是与RMP(other)几乎都在同一个数量级。该结果证明了即使毛干蛋白SAP分型检测的重现性存在差异,但是通过与样本的DNA序列分析比较,尤其是当RMP(self)较低时,呈现出了良好的个体区分能力。

Fig.5 All mutant SAPs identified in 12 samples
本文以毛干为研究对象,不仅分析了其蛋白质组成,而且针对SAP检验建立了相应的检测方法,并对重现性、准确性进行分析,最后评估了毛干SAP的个体识别能力。2 cm以上的毛干是案件现场可以获得的常见生物物证,本方法适用于实际应用需求。本方法与以往报道只使用突变型SAP位点[10-11,16-17]不同,不仅利用了突变型SAP,而且也利用了参考型SAP。在RMP计算时,参考型位点的加入和合并两个样本的检测结果,降低了RMP数值,从而提升了个体识别能力。角蛋白及相关蛋白被认为是毛干中的重要组成部分。通过GO分析发现,毛干蛋白质的组成成分中,最显著的是细胞外泌体类蛋白,其次才是角蛋白丝蛋白类,另外还有中间丝蛋白类、髓鞘蛋白类、细胞外基质蛋白类等各种不同种类的蛋白质。毛干的蛋白质组成是多样的,这种多样性是获得丰富SAP的前提。
毛干中相当大比例的SAP位点在角蛋白和角蛋白相关蛋白中[18]。将本方法与已报道的方法相比[10-11,16-17],SAP检出能力显著提高。不仅是因为本方法加入了参考型SAP,即使仅分析突变型SAP,在12个样本共检出了73个突变型SAP(有一个突变型位点在全部样本中都检出),多于Parker等[11]检出的33个和Mason等[10]检出的57个突变型SAP。分析本方法的优势有如下几点。a.离子液体对疏水性蛋白具有极强的溶解能力[19-20],可以提高蛋白质的提取,而超声过程的加入,则进一步增强了溶解能力,优于基于尿素提取法[11]和二硫苏糖醇(DDT)复合月桂酸钠(SDD)提取法[10];
与本研究组前期利用尿素裂解的结果[21]比较,离子液体前处理具有显著的优势,酶解后同样使用Nano-LC串联QE质谱仪检测,尿素法平均检出(937±262)个肽段,而离子液体法平均检出(2 027±385)个肽段。b.基于东亚人群的SAP数据使SAP位点的识别更具有针对性,同样有利于SAP位点的检出。c.受试个体全外测序中nsSNP结果与来源于公共数据的东亚人群SAP数据进行了整合,使部分不存在于公共数据库中的SAP位点得以被检出,如KRTAP1-1基因座上rs768488910位点(图4),并不存在于公共数据库中,但是通过对全外测序数据的整合,可以在样本F202A、F202B和样本M2A、M2B中被检出。
本文发现,同一个体两批次提取的毛干样本中,检出蛋白质和SAP数量存在差异,但这种差异与两次技术重复的差异接近,显示质谱采集方式可能是导致两次取样差异的重要影响因素。本文使用的质谱采集方式为数据依赖型采集(datadependent acquisition,DDA),每次采集TopN个质谱峰,具有一定的随机性[22-23],导致了两次取样检出的蛋白质及SAP存在一定差异。本文通过全外测序验证的办法,有效保证了检出位点的准确性,但也发现了半匹配的情况,即有一部分SNP位点为杂合型,SAP对应的SNP_pro仅检出其中一种分型,甚至还发现了部分SAP与SNP分型完全不一致的情况。其中半匹配的情况,除DDA方法的局限性以外,还可能是因为细胞内一条染色体转录与翻译活跃,而另一条染色体上等位基因转录或翻译收到抑制。而对于完全不一致的情况来说,原因比较复杂,至今仍未有确定的结论,这是今后需要深入开展研究的内容。
在个体识别应用方面,一个人基因组序列具有唯一性,而蛋白质组检测结果存在一定的差异性,本文首次提出以基因组为标准,通过蛋白质组SAP推导的nsSNP_pro与基因组中nsSNP匹配一致的位点并计算随机匹配概率,从而将蛋白质组与基因组有机联系起来。对蛋白质组和基因组来源于同一个人时,计算获得随机匹配概率最低(除1例因检出SAP位点过少以外)。该个体识别计算方法为后续法医毛干个体识别应用提出了一个有效的解决策略和应用场景,具有非常重要的应用价值。如现场有一根毛发,有5个嫌疑人可供排查时,本方法可以给出5个人相似性排序,可为锁定嫌疑人提供有力支撑。另外,未来还需在质谱检测方法上,针对毛干蛋白质组检测的特点,进一步改进毛干蛋白提取方式和加深蛋白质组检测覆盖度,以增加SAP检出数量。
本文建立了一个基于离子液体的毛干蛋白质组前处理及SAP质谱检测方法,并探索了个体识别分析流程,该方法具有毛发用量少、稳定可重复,检出SAP数量更多、针对东亚人群等优势,从随机匹配概率计算结果来看具有较好的个体识别能力。该方法有望成为法医DNA个体识别技术的有力补充,可以预期未来在法庭科学领域具有良好的应用前景。
附件见本文网络版(www.pibb.ac.cn或www.cnki.net):
PIBB_20210281_Table S1.pdf
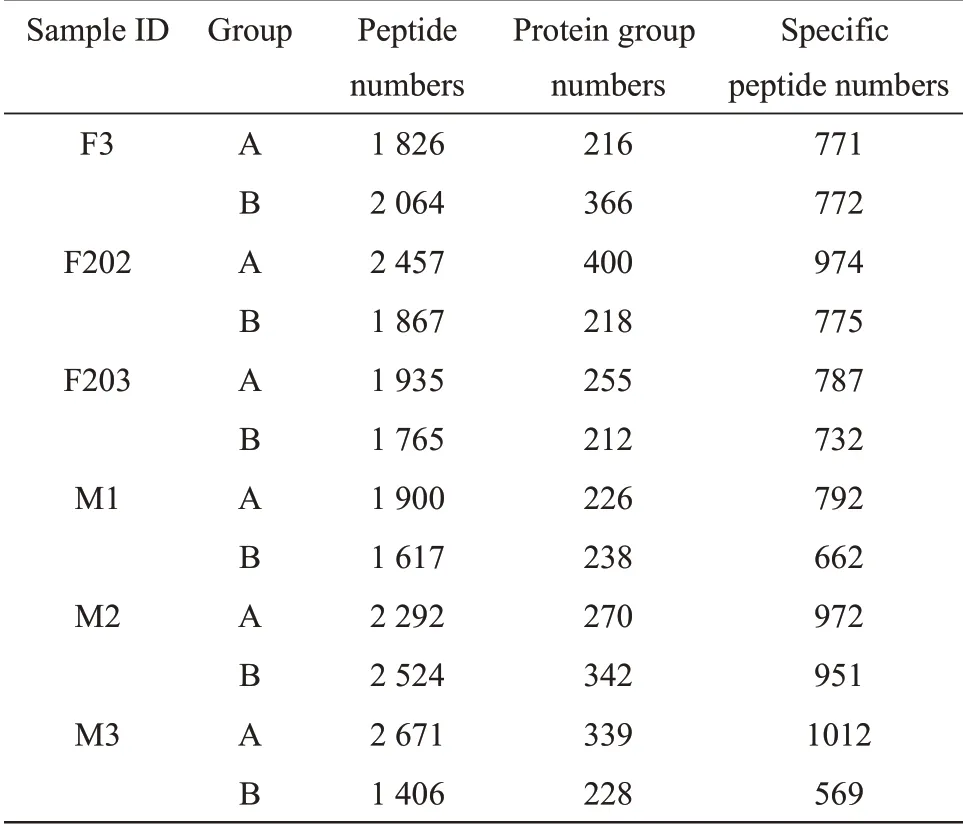
Table 1 The number of peptides,protein groups and unique peptides identified in 12 samples
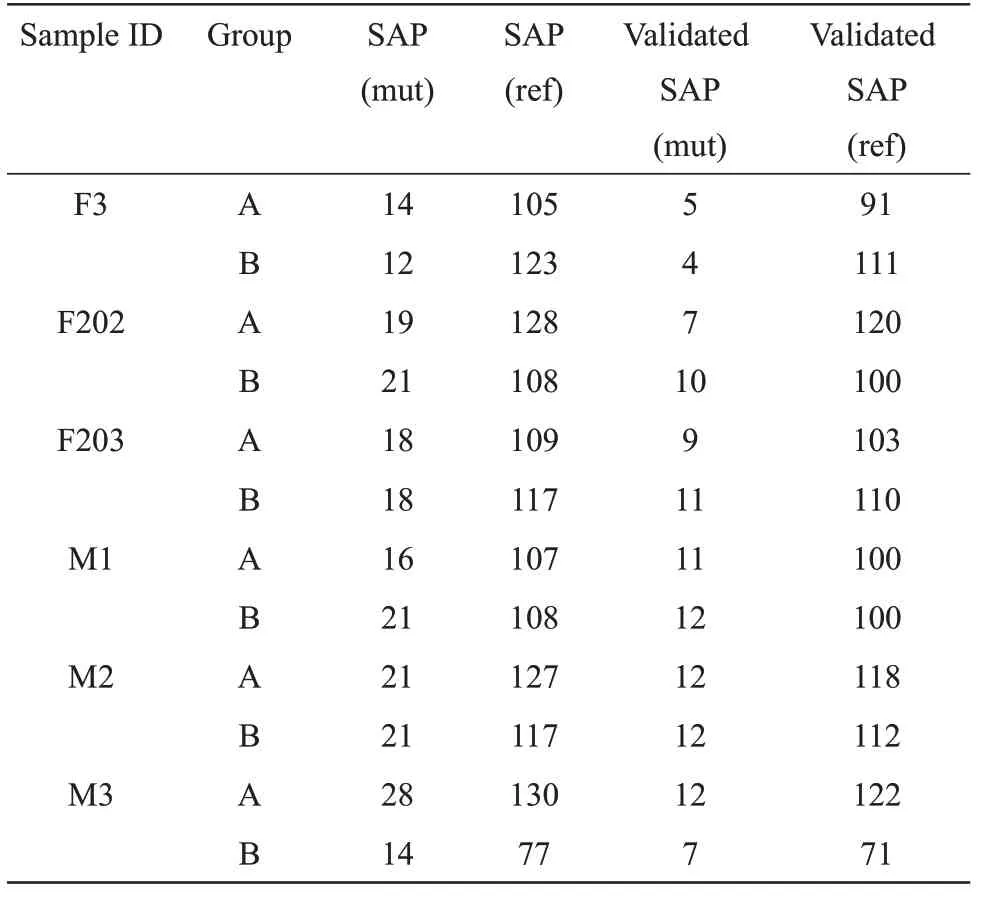
Table 2 The number of SAP identified in 12 samples
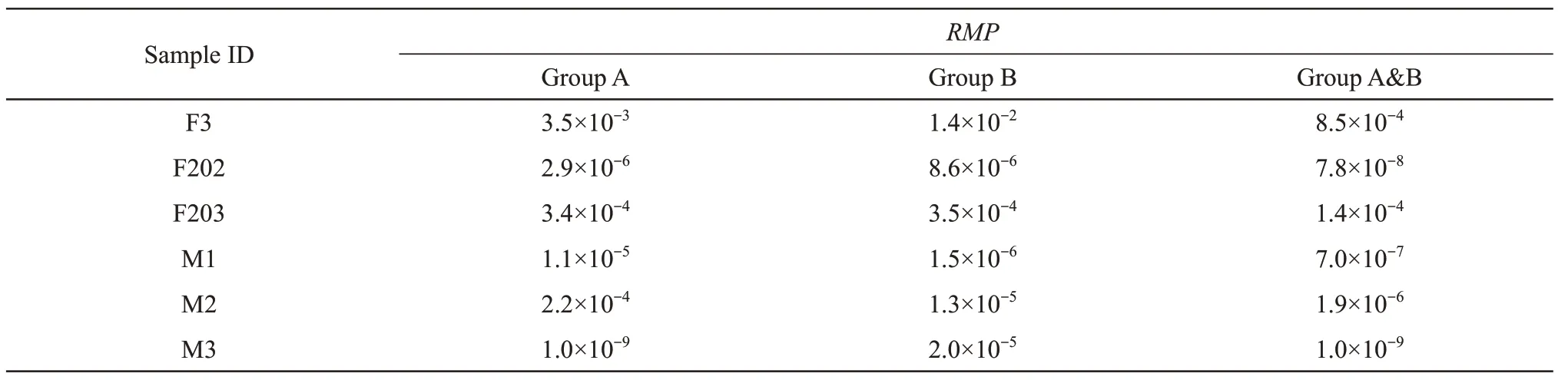
Table 3 RMP calculated by nsSNP_pro validated correctly by exome sequencing in 12 samples
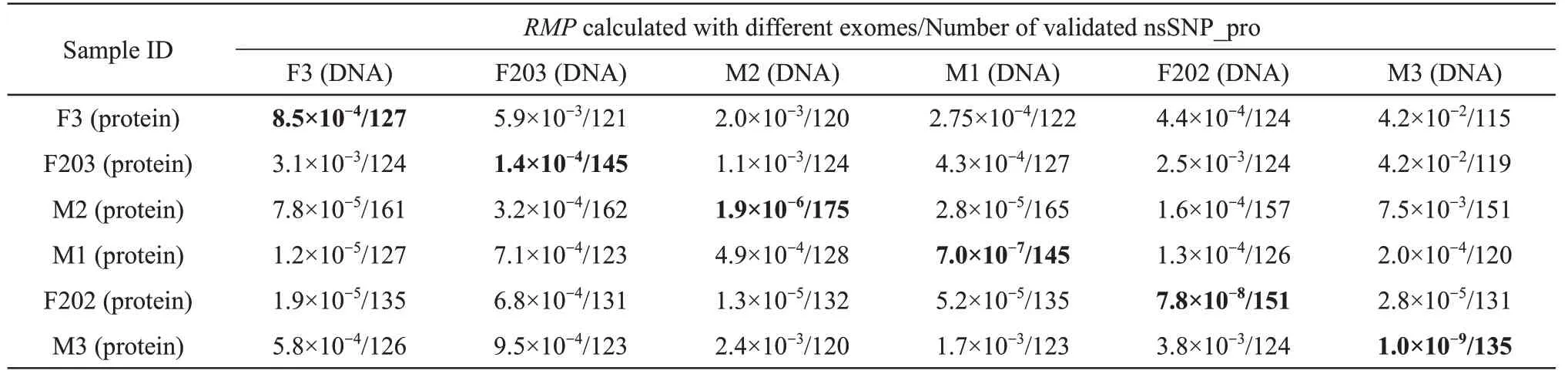
Table 4 Supposed RMP calculated by nsSNP_pro in accordance with different exomes in 12 samples
PIBB_20210281_Table S2.pdf
PIBB_20210281_Table S3.pdf
猜你喜欢 角蛋白分型位点 角蛋白家族基因及其与动物毛发性状关系的研究进展中国畜牧杂志(2022年1期)2022-11-06多环境下玉米保绿相关性状遗传位点的挖掘中国农业科学(2022年16期)2022-09-19PSORA:一种基于高通量测序的T-DNA插入位点分析方法中国农业科学(2022年15期)2022-08-09肺炎克雷伯菌RAPD基因分型及其与氨基糖苷类药敏分型对比研究中南药学(2022年2期)2022-03-30相信科学!DNA追凶是如何实现的?电脑报(2020年40期)2020-11-06兔毛角蛋白的制备及其在防晒化妆品中的应用研究科技风(2019年15期)2019-10-21羊毛角蛋白的溶解机制研究赤峰学院学报·自然科学版(2019年9期)2019-09-10CT三维及X线在股骨转子间骨折分型的可靠性中国社区医师(2019年1期)2019-06-26一种改进的多聚腺苷酸化位点提取方法电脑知识与技术(2018年19期)2018-11-01复杂分型面的分型技巧智能制造(2015年4期)2015-05-12推荐访问:东亚 多态性 氨基酸推荐文章
- [高考励志:倒计时冲刺语录] 高考倒计时励志语录
- 2018江苏高考成绩查询入口,点击进入:江苏高考2018成绩查询
- [英语阅读:细数英语中那些出口的汉语]带汉语的英语阅读视频
- 双语阅读:英文吐槽“变凉”的天气_喜剧中心吐槽大会2018
- 2018年福建高考成绩查询网址:http://www.eeafj.cn/:2018福建二建成绩查询
- 小学五年级下册语文阅读理解练习题五道_5年级下册语文书人教版
- 河北教育考试院2018高考查分_福建教育考试院网2018年高考查分系统
- 2018年山西省拟录用公务员公示 [2018年浙江瑞安市各级机关公务员拟录用人员公示(五)]
- 好舌头绕口令 [英语绕口令:挑战你的舌头]
- [2018福建高考成绩什么时候可以查询] 2018中级会计成绩查询