高粱淀粉合成酶基因家族鉴定与表达分析
来源:优秀文章 发布时间:2022-11-18 点击:
周光怡,李魁印,,李志友,姚茂星,王 睿,陈薇薇,吴传喜,任明见,2
(1.贵州大学,贵阳 550025;
2.国家小麦改良中心贵州分中心, 贵阳 550025;3.安顺学院农学院, 贵州 安顺 561000;4.桐梓县贵粱农业发展有限责任公司, 贵州 遵义 563200)
高粱[Sorghumbicolor(L.) Moench]是世界第五大粮食作物,仅次于玉米、小麦、水稻和大麦。籽粒高粱按总淀粉的含量以及支链淀粉所占的比例可分为粳高粱和糯高粱,支链淀粉占比高的糯高粱是贵州酱香型白酒的主要酿酒原料之一,总淀粉含量与支链淀粉所占比例对酱香型白酒的风味、品质有着决定性作用[1]。淀粉以淀粉粒的形式存在于胚乳中,是禾谷类作物产量、品质形成的重要组分[2-3]。淀粉的生物合成包括两种形式,一种是在光合组织叶绿体中进行瞬时淀粉的合成,另一种是在造粉体中完成贮藏淀粉的合成[4]。
Cao H等[5]根据淀粉合成酶基因的保守序列进行分类,认为禾谷类作物胚乳中至少含有5种SS同工酶,即GBSS、SSⅠ、SSⅡ、SSⅢ和SSⅣ,根据理化性质可分为颗粒结合型淀粉合成酶(Granule-bound starch synthase,GBSS)和可溶性淀粉合成酶(Soluble starch synthase,SSS)两类。李祥栋等[2]研究发现,GBSS基因直接参与胚乳中直链淀粉的生物合成途径。此外,淀粉的生物合成代谢还需要多种酶及其同工酶的相互调控,主要包括二磷酸腺苷葡萄糖焦磷酸化酶(ADP-glucose pyrophosphorylase,AGPase)、淀粉分支酶(Starch branching enzyme,SBE)、淀粉去分支酶(Starch debranching enzyme,DBE)和质体淀粉磷酸化酶(Plastidial starch phosphorylase,Pho)等[6]。
前人在水稻、玉米、谷子等物种中都鉴定出淀粉合成酶基因家族,并对其进行详细报道,但对高粱淀粉合成酶基因家族的相关研究较少。本研究通过生物信息学方法,对高粱淀粉合成酶基因家族的理化性质、系统进化树、蛋白结构和顺式作用元件等方面进行研究,并通过转录组数据对该家族基因进行表达模式分析,为高粱淀粉合成酶分子水平上的进一步研究和贵州酒用糯高粱的分子选择育种提供一定的参考依据。
1.1 高粱淀粉合成酶基因家族成员鉴定
以水稻和玉米的淀粉合成酶家族基因序列作为参考,在NCBI(https://www.ncbi.nlm.nih.gov/)和Phytozome(https://phytozome-next.jgi.doe.gov/)两个数据库进行BlastP比对[7],根据NCBI的注释功能筛选出11个高粱淀粉合成酶基因家族成员。
1.2 高粱淀粉合成酶基因家族蛋白质理化性质分析
将蛋白序列提交至在线蛋白质分析网站ExPASy(https://web.expasy.org/protparam/),对该基因家族做蛋白长度、分子量、等电点、不稳定系数、脂溶系数以及蛋白亲疏水性的预测[8],采用Softberry分析软件(http://www.softberry.com/)对11个基因进行亚细胞定位。
1.3 高粱淀粉合成酶基因家族系统进化树及聚类分析
利用MAGA 11软件对高粱淀粉合成酶家族基因的蛋白序列进行Clustal W比对[9],将比对结果采用最大似然法(Maximum likelihood,ML)构建系统进化树,通过1 000次校验来增加分析的可靠性,进行系统进化树分析,导出NWK文件,用在线进化树美化软件EVOLVIEW(https://www.evolgenius.info/evolview-v 2)对进化树进行美化[10]。
1.4 高粱淀粉合成酶家族基因的染色体定位分析
根据已比对筛选出的高粱淀粉合成酶家族的基因号和基因组注释文件,找到该家族成员的染色体位点,并利用在线绘图软件MG 2 C(http://mg2c.iask.in/mg2c_v2.1/)绘制高粱淀粉合成酶家族基因的染色体分布图[11]。
1.5 高粱淀粉合成酶基因家族蛋白二级结构分析及三级结构预测
利用SOPMA(https://npsa-prabi.ibcp.fr/)对高粱淀粉合成酶基因家族蛋白质的二级结构进行预测分析[12];
提交家族基因的蛋白序列至蛋白质三级结构在线预测网站SwissModel(https://swissmodel.expasy.org/interactive)构建模型[13],对SOPMA预测的二级结构的准确性进行检验。
1.6 高粱淀粉合成酶基因结构及基因保守motif分析
利用GSDS 2.0在线分析网站(http://gsds.gao-lab.org/)绘制基因结构图[14];
利用在线工具MEME(https://meme-suite.org/meme/org)对高粱淀粉合成酶基因家族保守基序进行分析[15],设置motif数为8,其余为默认参数。
1.7 高粱淀粉合成酶基因家族启动子顺式元件分析
利用TBtools软件提取起始密码子上游2 000 bp的基因序列[16],上传至PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/),进行启动子顺式作用元件分析[17],在TBtools软件进行可视化绘图。
1.8 高粱淀粉合成酶在不同发育阶段中的表达模式分析
通过ArrayExpress数据库[18](https://www.ebi.ac.uk/arrayexpress/)查询已公布的高粱转录组数据,分析了Wang B等[19]在Btx 623不同发育阶段取样的转录组数据(数据代码:E-MTAB-5956),包括萌发后14 d的根、茎、叶和幼苗,授粉后20 d的胚、胚乳和果实,9~10周阶段的花粉和3种不同长度的花序时期。数据整理后用在线绘图工具BioLadder(https://www.bioladder.cn/)绘制表达模式热图。
2.1 高粱淀粉合成酶基因家族成员鉴定
为全面了解高粱淀粉合成酶的进化历程及其在高粱淀粉合成中的重要性,为高粱淀粉合成酶基因家族进行全面分析。以水稻和玉米淀粉合成酶基因家族为参考,经过对比与筛选,在高粱上遴选出11个淀粉合成酶基因,参考水稻淀粉合成酶基因家族名称及NCBI上对应基因的描述,发现高粱该家族中除了SSⅠ和SSⅤ没有同工酶外,GBSS、SSⅢ和SSⅣ都有两种同工酶,SSⅡ有三种同工酶,家族成员基因命名及基因基本信息如表1所示。

表1 高粱淀粉合成酶基因家族基本信息Table 1 Information of sorghum starch synthase gene family
2.2 高粱淀粉合成酶家族基因蛋白质理化性质分析
对以上11个淀粉合成酶家族基因所编码的蛋白进行理化性质分析,结果(表2)表明,氨基酸残基数目在607(SbGBSSⅡ)~1 680(SbSSⅢa)之间,分子量在66 073.95(SbGBSSⅠ)~190 017.22(SbSSⅢa)之间,可见颗粒结合型淀粉合成酶(SbGBSSⅠ、SbGBSSⅡ)的序列长度与分子量明显小于可溶性淀粉合成酶(SbSSⅠ~SbSSⅤ)。在可溶性淀粉合成酶中,SbSSⅢ两个基因编码的蛋白(XP_021320945.1和XP_021318466.1)长度和分子量都显著大于其他9个基因。家族基因的蛋白质等电点介于5.01(SbSSⅢa)和6.37(SbGBSSⅠ)之间,说明高粱淀粉合成酶基因家族所编码的蛋白均为酸性蛋白;
蛋白不稳定系数在29.02(SbGBSSⅡ)~53.58(SbSSⅢa)之间,除GBSS和SSⅠ两个亚家族中的三个蛋白(SbGBSSⅠ、SbGBSSⅡ、SbSSⅠ)为稳定蛋白外,其余的8个蛋白不稳定系数均大于40,蛋白稳定性较差;
基因家族蛋白的脂溶系数在73.21(SbSSⅢb)~91.01(SbSSⅤ)之间,亲水系数平均值均小于0,表明高粱淀粉合成酶基因家族蛋白为亲水蛋白,且流动性较好。对已知的高粱淀粉合成酶基因家族成员进行亚细胞定位,结果表明,SbGBSS Ⅰ、SbGBSS Ⅱ、SbSS Ⅲ a、SbSS Ⅲ b和SbSS Ⅳ a等5个蛋白定位在细胞外,SbSS Ⅰ、SbSS Ⅱ a、SbSS Ⅱ b、SbSSⅡc、SbSSⅣb和SbSSⅤ等6个蛋白定位在叶绿体上。

表2 高粱淀粉合成酶蛋白质理化性质Table 2 Physicochemical properties of sorghum starch synthase protein
2.3 淀粉合成酶系统进化树分析
为了探究高粱淀粉合成酶基因家族的进化关系,对部分单子叶禾本科植物和双子叶模式植物的淀粉合成酶家族基因进行聚类分析,包括9个玉米淀粉合成酶基因、11个水稻淀粉合成酶基因、9个谷子淀粉合成酶基因和6个拟南芥淀粉合成酶基因。各物种间的淀粉合成酶的进化关系如图1所示,46个不同物种的淀粉合成酶基因被划分为5个分支,各同工酶间相互存在同源性,其中,GBSSⅠ与GBSSⅡ、SSⅣ与SSⅤ的进化距离较近,基因都可以聚类同一分支;
再者,ZmGBSSⅠ与SbGBSSⅠ、ZmGBSSⅡ与SbGBSSⅡ、SiSSⅡa与SbSSⅡa、ZmSSⅡb与SbSSⅡb、ZmSSⅡc与SbSSⅡc、ZmSSⅢa与SbSSⅢa、ZmSSⅢb与SbSSⅢb、OsSSⅣb与SbSSⅣb互为直系同源基因,可以看出,玉米与高粱的淀粉合成酶基因在进化过程中的同源关系最近,其次是谷子和水稻,而拟南芥与单子叶植物的淀粉合成酶家族基因之间的进化距离较远。

图1 淀粉合成酶基因家族的系统进化树分析Fig.1 Phylogenetic tree analysis of the starch synthase gene family

图2 高粱淀粉合成酶基因的染色体分布Fig.2 Chromosome distribution of sorghum starch synthase gene
2.4 高粱淀粉合成酶基因家族染色体定位分析
通过对高粱淀粉合成酶11个基因进行染色体定位分析,发现各基因并未均匀分布在高粱的10条染色体上,在Chr 3、Chr 5、Chr 8染色体上均未见该基因家族成员基因的分布,在其余7条染色体中分布也不均匀(图2),在Chr 4、Chr 10染色体上各分布3个基因,Chr 1、Chr 2、Chr 6、Chr 7和Chr 9染色体上各分布1个基因。该家族基因均分布在各染色体的两端,各同工酶基因并没有映射到同一条染色体上,而是散布在各染色体中。
2.5 高粱淀粉合成酶基因家族蛋白二
级结构分析及三级结构预测
高粱淀粉合成酶基因家族蛋白的二级结构以α-螺旋和无规则卷曲为主,二者在各蛋白质中所占核苷酸数量均大于70%,延伸链与β-转角占比较少(表3),可知高粱淀粉合成酶基因家族蛋白是由α-螺旋和无规则卷曲支撑,而延伸链和β-转角则散布在整个蛋白中。
利用在线网站SwissModel对基因蛋白模型进行预测,图3为高粱淀粉合成酶基因家族的蛋白三级结构预测结果,可以直观地看出,11个蛋白均以α-螺旋和无规则卷曲为主要结构元件,与其二级结构结果吻合。家族中11个蛋白的结构相似,SSⅠ、SSⅡ中4个基因的蛋白三级结构相似度较高,GBSS、SSⅢ、SSⅣ和SSⅤ中的7个基因的蛋白三级结构相似度较高。
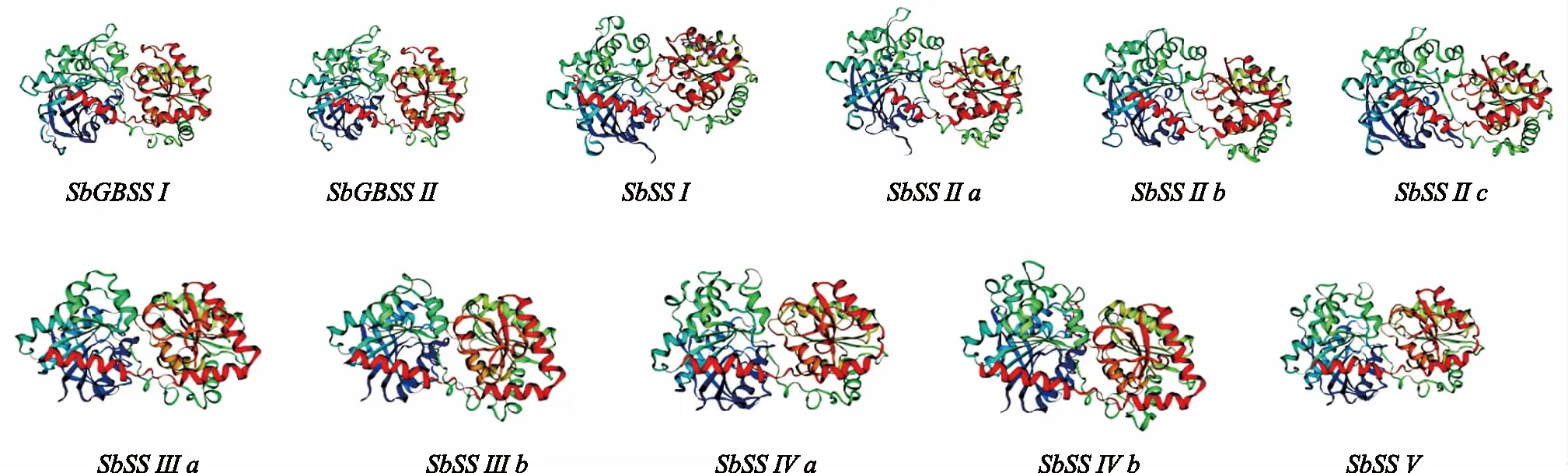
图3 高粱淀粉合成酶基因蛋白三级结构预测Fig.3 Prediction of protein tertiary structure of sorghum starch synthase gene

图4 高粱淀粉合成酶基因结构Fig.4 Gene structure of sorghum starch synthase

表3 高粱淀粉合成酶基因蛋白二级结构预测Table 3 Prediction of protein secondary structure of sorghum starch synthase gene
2.6 高粱淀粉合成酶基因结构及保守基序分析
高粱淀粉合成酶基因家族的基因结构如图4所示,除了SbSSⅡc没有下游非编码区,其余每个基因均有长短不一的上下游非编码区。CDS(Coding sequence)是指可以被翻译成蛋白质的编码序列区域,即外显子;
Intorn是编码区域间没有编码作用的序列,即内含子。大部分基因的外显子数目在8~19个之间,SbGBSS、SbSSⅡ和SbSSⅢ中的基因成员分别有13、8个和16个外显子,可以看出各同工酶基因在外显子数目、长度和分布位置上在一定程度相似。

图5 高粱淀粉合成酶家族保守基序Fig.5 Conserved motifs of sorghum starch synthase family
对高粱淀粉合成酶家族基因的蛋白质序列进行保守基序分析(图5),结果显示,高粱淀粉合成酶基因家族共有8种保守基序,保守基序的氨基酸残基数在29~40之间。其中4个保守基序(motif 1、motif 2、motif 5和motif 8)在11个基因成员蛋白质序列中都有分布,motif 4分布在GBSS、SSⅠ和SSⅡ中,motif 7分布在SSⅢ、SSⅣ和SSⅤ中,motif 6在除GBSS外的其他基因中都有分布,motif 3在除SbSSⅣa和SbSSⅤ外的基因中都有分布。从motif的分布位置来看,高粱淀粉合成酶的基序特征与系统进化树分析结果吻合,各同工酶间motif位置分布情况几乎一致。

图6 高粱淀粉合成酶启动子顺式作用元件Fig.6 Sorghum starch synthase promoter cis-acting element
2.7 高粱淀粉合成酶基因家族启动子顺式元件分析
为更好地探索高粱淀粉合成酶基因调控表达,提取高粱淀粉合成酶基因家族起始密码子上游2 000 bp的序列,对这部分序列进行顺式作用元件分析,发现该区域存在35种顺式元件,主要分为三类,包括光响应元件、激素响应元件和胁迫响应元件,如图6所示。在SbGBSSⅡ和SbSSⅢa的启动子区域发现与淀粉合成直接相关的顺式调节元件GCN 4_motif,在SbSSⅢa发现参与昼夜节律控制的顺式调节元件circadian;
光响应元件在高粱淀粉合成酶基因家族中广泛分布,SbSSⅡc存在26个光响应元件,SbSSⅣb只有6个。家族中还存在一定数量的激素响应元件,包括参与脱落酸反应的顺式作用元件ABRE、调控赤霉素反应元件P-box和GARE-motif、调控生长素反应元件TGA-element等,另外在可溶性淀粉合成酶SSI~SSIV中都发现了参与低温反应的顺式作用元件LTR,但在颗粒型淀粉合成酶GBSS两个基因中并未发现该元件。
2.8 高粱淀粉合成酶家族基因表达模式分析
在高粱不同发育阶段的转录组数据中筛选出淀粉合成酶基因家族的基因表达数据,并绘制热图进行分析,该家族基因在高粱不同发育阶段中都有表达,但FPKM值差异较大。如图7所示,SbGBSSⅠ、SbSSⅡc和SbSSⅠ在授粉20 d的胚、胚乳和果实中高表达,其中SbGBSSⅠ在胚乳中的FPKM值最大,平均为1 579.2;
SbSSⅠ、SbGBSSⅡ和SbSSⅢb在开花14 d的根、茎、叶和幼苗中高表达,其中在叶片中的FPKM值最大;
SbSSⅠ、SbGBSSⅡ、SbSSⅢb在花序展开的三个时期高表达,SbSSⅡc和SbSSⅣa两个基因在不同时期不同部位的FPKM值都较低。

图7 高粱淀粉合成酶基因表达热图Fig.7 Heat map of sorghum starch synthase gene expression
对高粱基因组进行对比、筛选,鉴定出了11个高粱淀粉合成酶基因家族成员,与玉米[20]、水稻[21]、谷子[22]、苋菜[23]等植物的鉴定结果一致。通过对高粱淀粉合成酶基因家族理化性质的分析,发现11个高粱淀粉合成酶基因家族所编码的蛋白均为酸性蛋白,且均是亲水性蛋白,整体稳定性较差,但流动性较好。对高粱、玉米、水稻、谷子和拟南芥的淀粉合成酶基因进行系统进化树分析,发现以上5个物种的同工酶可以聚类在一起,说明植物淀粉合成酶基因在进化过程中高度保守。其次,进化树聚类分成两个分支,一个分支包括GBSS、SSⅢ、SSⅣ和SSⅤ,另一个分支包括SSⅠ和SSⅡ,推测淀粉合成酶家族基因可能是来自于2个祖先基因。高粱淀粉合成酶基因家族与玉米的亲缘关系最近,谷子次之,而拟南芥与单子叶植物淀粉合成酶家族成员间的进化距离较远,推测淀粉合成酶在植物进化过程中的某一时期出现了单双子叶之分。
蛋白质复杂的组成和结构是其多样性生物学功能的基础,不同蛋白结构决定着高粱淀粉合成酶功能的多样性[24]。对家族成员的蛋白二级结构和三级结构进行了预测和分析,结果都与系统进化树的结果一致,通过基因结构与motif分析,再次验证了各同工酶之间的进化关系。顺式作用元件本身不编码蛋白,但它们参与基因表达的调控,对高粱淀粉合成酶家族基因启动子区域的顺式作用元件进行分析,发现该区域存在大量光响应元件和激素响应元件,但高粱淀粉合成酶与脱落酸之间存在的关系并不清晰,有待进一步研究。在除颗粒结合型淀粉合成酶外的9个基因中均发现LTR元件(参与低温反应的顺式作用元件),推测可溶性淀粉合成酶可能参与了高粱低温介导的信号通路,在高粱耐低温机制中起着一定的调节作用[25]。
对各基因在不同部位间的FPKM值绘制热图分析,发现高粱淀粉合成酶基因主要在叶片和胚乳中高表达,这与植物淀粉合成场所和贮藏部位吻合。李晓兵[20]在玉米淀粉合成酶基因家族的时空表达分析中认为,玉米淀粉合成酶基因在授粉后随时间推移整体呈上升趋势,在叶片中的相对表达量最大。本研究结果与之相似。
淀粉合成酶在谷物胚乳淀粉生物合成过程中发挥关键作用,在与其他淀粉合成相关酶的相互作用下,进而影响谷物的质量与产量[6]。本研究对高粱淀粉合成酶基因家族成员的系统进化树、亚细胞定位、蛋白结构和表达模式等进行了较为全面的分析,以期为高粱淀粉合成酶分子水平上的进一步研究和贵州酒用糯高粱的分子选择育种提供一定的参考依据。
猜你喜欢 元件高粱淀粉 高粱红了青年文学家(2022年1期)2022-03-11糯米和大米阅读(科学探秘)(2021年11期)2021-03-10为什么粥凉了更稠祝您健康·文摘版(2021年3期)2021-03-09淀粉裹多了肉会老饮食与健康·下旬刊(2018年3期)2018-04-11野高粱高中生学习·高二版(2017年7期)2017-09-23雨后诗潮(2017年2期)2017-03-16如何读懂色环电阻中学生数理化·中考版(2016年7期)2016-12-07非线性元件的处理中学物理·高中(2016年8期)2016-08-08碘遇淀粉一定变蓝色吗中学化学(2014年11期)2015-01-20上海风云“东邪”谁属现代计算机(2009年9期)2009-12-02推荐访问:高粱 淀粉 基因